Pequeños ARN
El arte de evadir al sistema inmune
Hasta hace un par de décadas, el ácido ribonucleico (ARN) era considerado un simple intermediario entre el ADN, la unidad que contiene toda la información genética, y las proteínas. Posteriormente, el descubrimiento de que unas moléculas llamadas "pequeños ARN" controlan gran parte del comportamiento genético fue considerado el principal logro del año 2002, según los científicos de la American Association for the Advancement of Science (AAAS), editores de la prestigiosa revista 'Science'.
En estudios publicados durante 2004, se encontró que el citomegalovirus de ratón es capaz de mutar para evadir las células asesinas naturales. Éstas son elementos importantes del sistema inmune innato que ataca primero a las infecciones. Este componente más generalizado entra en acción rápidamente para detener la evolución infecciosa. En el proceso, le otorga un tiempo valioso a la segunda y más específica línea de defensa del sistema inmunológico, conocida como inmunidad adquirida, que debe adaptarse y proliferar para atacar a un virus o a un microorganismo invasor particular. Estudiando la versión más reciente del juego de las escondidas entre patógenos y los huéspedes que infectan, investigadores han encontrado que el citomegalovirus parece camuflarse con un dispositivo de silenciamiento genético recientemente descubierto, que utiliza para evitar ser detectado y destruido por las células inmunes. En personas, plantas y gusanos, centenares de moléculas minúsculas de ARN pueden silenciar genes específicos interfiriendo los ARN mensajeros más grandes. Esa interferencia evita que los mensajeros hagan proteínas. Los científicos no saben qué genes son silenciados por los micro ARN en las personas, pero las nuevas publicaciones refuerzan la creciente evidencia de que las pequeñas moléculas de ARN pueden desempeñar funciones importantes no sólo en células humanas normales sino también en células infectadas.
En concreto, el conocimiento popular y científicamente aceptado es que el complejo sistema de generación de ARN pequeños fue diseñado para que representara una defensa particularmente específica de las células contra los virus y otros patógenos. Los estudios ahora demuestran que un virus también puede adaptarlo para evadir la inmunorrespuesta.
Micro ARN y grandes virus
Aunque la mayoría de las infecciones por citomegalovirus humano (CMV) son asintomáticas, es un importante patógeno en los individuos inmunodeprimidos, como los pacientes con SIDA y aquellos que reciben terapia inmunosupresora después de un trasplante de órgano o de médula ósea. Además, la infección por CMV es una de las principales causas de defectos de nacimiento y se ha implicado en la aceleración de enfermedades vasculares como la aterosclerosis. Al igual que el contagio con otros herpesvirus, la infección primaria es seguida por una que persiste durante toda la vida o por un estado latente del virus en el hospedador. La clave de la latencia es que las células infectadas eviten el reconocimiento inmunológico y permanezcan “sanas” durante la producción de proteínas virales que pueden ser tóxicas para la célula. Estudios recientes han arrojado luz sobre cómo el CMV puede realizar estas tareas utilizando ARN expresados por el mismo virus.
Los herpesvirus humanos, incluidos los citomegalovirus, codifican múltiples micro ARN (miARN), con 21 a 24 nucleótidos de largo. Estos ARN (que no son traducidos a proteínas) regulan la expresión de genes específicos dirigidos directamente por el ARN mensajero (ARNm), por lo tanto median su degradación o la inhibición de su traducción. Los miARN celulares regulan la expresión génica en los procesos de desarrollo y recientemente se ha comunicado que estarían asociados con tumores malignos. Sin embargo, la función y los objetivos de los miARN virales son en gran medida desconocidos. Sólo unas pocas bases de este mensajero se unen a su objetivo, lo que hace difícil identificar el propósito final a través de búsquedas computacionales.
Stern-Ginossar y colaboradores han podido superar estos obstáculos mediante una nueva herramienta bioinformática utilizada para identificar el complejo mayor de histocompatibilidad (o MHC, acrónimo del inglés para major histocompatibility complex) clase I relacionado con la cadena B (MICB) del ARNm objetivo de un miARN codificado por el CMV. MICB es un ligando celular para la activación del receptor NKG2D, el cual se expresa en algunas células asesinas naturales, en células T gama/delta y en linfocitos T CD8 +. Durante el estrés celular, como el causado por la infección viral, MICB es inducido por las células asesinas naturales y las células T, lo que puede conducir a la muerte de las unidades infectadas. Por lo tanto, el bloqueo de este proceso probablemente beneficiaría al virus. De hecho, el grupo de Stern-Ginossar demostró que las células infectadas por CMV que han sido diseñadas sin miARN (miR-UL112-1) son más susceptibles de ser eliminadas en un modelo dependiente de NKG2D por las células asesinas naturales. El tipo de CMV que codifica la proteína UL16, no fue capaz de modificar su expresión, tampoco tuvo protección contra la detección de células infectadas por las células asesinas naturales, ni secuestró a MICB en el medio intracelular lo que le impidió alcanzar la superficie celular.
¿Por qué el virus tiene dos mecanismos diferentes para lograr el mismo objetivo? No está claro, especialmente respecto a la estrecha relación inducida durante la infección viral entre el receptor activador NKG2D y el ligando MICA. UL16 es más eficiente que el miARN en la regulación a la baja de MICB en la superficie celular, de modo que tal vez la proteína viral es esencial para la evasión del sistema inmune durante la replicación aguda del virus, cuando muchos antígenos virales están presentes en la célula y la respuesta inmunológica del hospedador es probablemente más vigorosa. Asimismo, el miARN puede adecuarse mejor que UL16 en la regulación a largo plazo de MICB durante la infección persistente o latente por CMV. Utilizando un miARN en lugar de una proteína, el virus puede estar en mejores condiciones de "ocultarse" del sistema inmune.
Los miembros de mi laboratorio y yo recientemente hemos descrito otra función de los mismos miARN investigados por Stern-Ginossar y colaboradores. De manera exitosa, la persistencia de la infección depende del mantenimiento de la viabilidad celular a pesar de la producción de proteínas virales tóxicas. Una forma en que CMV puede limitar la producción de proteínas virales es mediante la restricción de la replicación viral. Hemos observado que miR-UL112-1 regula hacia abajo la expresión génica de CMV implicada en su propio proceso de replicación, en parte por la orientación del ARNm del huésped (codificación de una proteína llamada inmediata-temprana 72) que regula la transcripción de los genes virales necesarios para la replicación aguda.
El CMV también posee mecanismos que se oponen a la apoptosis de las células infectadas. Éstos codifican proteínas, tales como el inhibidor de la apoptosis que se localiza en la mitocondria y un inhibidor de la caspasa 8 inductora de la apoptosis. Reeves y colaboradores acaban de describir un nuevo mecanismo antiapoptótico mediado por el ARN B2.7 del CMV. La función de este ARN transcrito abundantemente durante la primera fase de la replicación vírica ha dejado perplejos a los investigadores desde su descripción original. El grupo de trabajo de Reeves demostró que el ARN B2.7 se une a los componentes del complejo I de la cadena respiratoria mitocondrial, estabilizando el complejo, como garantía de que permanecerá vinculado a la membrana mitocondrial interna y, por tanto, prevendrá la apoptosis. También en este caso (es decir, la inhibición de la apoptosis), el miARN puede tener una ventaja sobre sus homólogos al reducir la posibilidad de reconocimiento inmune.
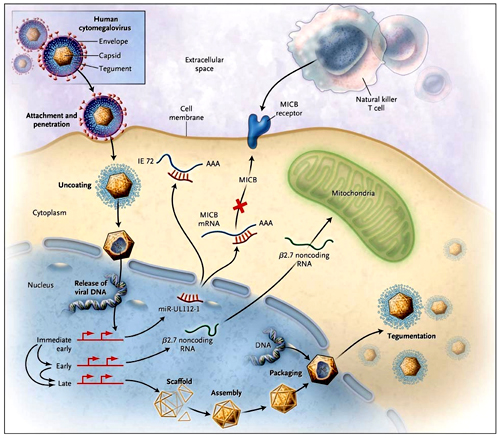
Para una eficiente replicación viral, el citomegalovirus humano (CMV) debe mantener la viabilidad celular evitando el sistema inmunológico del hospedador y los mecanismos de defensa innatos como la apoptosis. Tres recientes estudios indican que el CMV lo hace manipulando las respuestas virales y celulares a través de ARN no codificadores. La actividad del miARN (miR) UL112-1 reduce la expresión de los principales complejos de histocompatibilidad de clase I relacionados con la cadena B (MICB), concediendo protección contra el ataque de las células asesinas naturales, y atenuando la replicación aguda mediante ARNm que codifican para la proteína viral inmediata-temprana 72 (IE72). El ARN B2.7 bloquea la apoptosis por estabilización del complejo I de la cadena respiratoria mitocondrial. AAA señala la secuencia de poliadenilación.
Estos estudios sugieren que para una infección viral el CMV utiliza un ARN no codificante que permite optimizar el entorno celular. La figura 1 ilustra los mecanismos de miR-UL112-1 y la función del ARN B2.7 durante el ciclo de vida del CMV. Estas investigaciones también plantean la posibilidad de nuevos enfoques para la lucha contra la infección. Los inhibidores de las pequeñas moléculas de miARN son muy eficaces y específicos en cultivos de tejidos. La eficacia o efectos tóxicos de éstos en los animales se desconocen. La orientación de miR- UL112-1 con el uso de tales inhibidores podría conducir al virus hacia la replicación aguda mientras que la interrupción de la inhibición de la expresión de MICB resultaría en la posible remoción inmunológica del virus. Una segunda opción sería imitar la expresión de miR-UL112-1 con la utilización de miARN sintéticos en un intento de atenuar la replicación de la fase aguda del virus. A diferencia de muchos agentes antivirales que artificialmente bloquean los procesos virales, la liberación endógena de miARN virales podría aprovechar el propio mecanismo viral para reprimir la replicación. Por último, pequeños ARN dirigidos contra ARN B2.7 podrían utilizarse para intervenir su escisión. La reducción del ARN B2.7 daría lugar a la apoptosis de la célula infectada y quizás a la limpieza de los reservorios virales.
Fuente bibliográfica
Small RNAs and Large DNA Viruses
Jay A. Nelson, Ph.D.
Vaccine and Gene Therapy Institute, Oregon Health and Science University, Beaverton, USA.
N Engl J Med. 2007 Dec 20;357(25):2630-2