Los blancos moleculares de rapamicina
La rapamicina (sirolimus) se identificó a principios de los años setenta como un compuesto macrólido con propiedades antimicóticas. Es producido por una bacteria, Streptomyces hygroscopicus, cultivada a partir de una muestra de suelo aislada en Rapa Nui. El interés en la rapamicina se intensificó cuando se descubrió que atenuaba la proliferación de células de mamíferos, incluyendo las del cáncer y células inmunitarias, lo que ha cautivado científicos y clínicos por más de 40 años.
Crecimiento celular
El premio Albert Lasker de Investigación Médica Básica del año 2017, anunciado el 6 de septiembre, reconoció a Michael N. Hall de la Universidad de Basilea por su descubrimiento del blanco de la rapamicina (TOR), una enzima sensor de nutrientes que regula la actividad celular y crecimiento del organismo en casi todas las especies eucariontes. Debido a su capacidad para inhibir el crecimiento celular, el producto natural rapamicina ha cautivado a científicos y clínicos por más de 40 años.
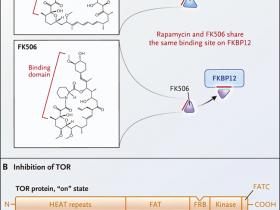
La supresión de la activación de las células T por rapamicina, así como su similitud a otro inmunosupresor derivado de estreptomices, el macrolido FK506 (también conocido como tacrolimus), llevó a los primeros descubrimientos en su mecanismo de acción. Ambos inmunosupresores se unen e inhiben una peptidil prolil isomerasa llamada proteína de unión a FK506 (FKBP12) a través de motivos comunes a las dos moléculas (figura 1A).
Aunque la supresión de la activación de las células T mediante rapamicina y FK506 fue dependiente de su unión a FKBP12, no estaba claro por qué la rapamicina y el FK506 tuvieron efectos distintos en la señalización de las células T y podrían antagonizar sus acciones. La disección de estas propiedades por Stuart L. Schreiber y colaboradores (Science 1991; 251: 283-7) condujo a la hipótesis de que era un complejo proteico FKBP12-rapamicina que provoca la respuesta de crecimiento supresivo a través de un objetivo desconocido, lanzando una carrera por el descubrimiento del blanco de la rapamicina. La sorprendente conservación evolutiva del mecanismo de acción de rapamicina resultaría fundamental.
Michael Hall y sus colegas emprendieron una investigación genética para detectar mutantes resistentes a la rapamicina en la levadura Saccharomyces cerevisiae (Cell 1993; 73: 585-96). La rapamicina bloquea la entrada al ciclo celular en S. cerevisiae. Esta propiedad de la rapamicina permitió a Hall y colegas identificar a 18 levaduras mutantes resistentes a la rapamicina que eran capaces de formar colonias en placas de cultivo con rapamicina. Quince de estos mutantes tenían una variante "etiológica" en la levadura ortóloga de FKBP12 (el gen se llama FPR1). Cruces genéticos revelaron que los otros tres mutantes no eran alélicos con FPR1(es decir, no tenían mutaciones en este gen).
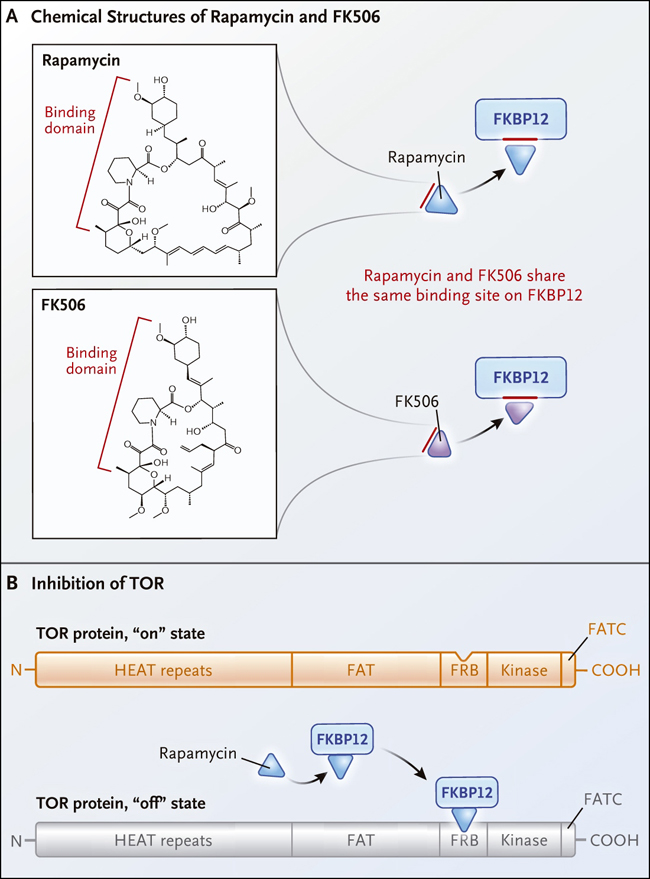
Figura 1. El mecanismo de rapamicina.
Los compuestos inmunosupresores rapamicina (sirolimus) y FK506 (tacrolimus) tienen una fracción química común que se une a la proteína FKBP12 (panel A). Para inhibir el blanco de las proteínas de rapamicina (TOR) (panel B), la rapamicina debe unirse primero al FKBP12, con el complejo proteico - fármaco y luego unirse al dominio FKBP - rapamicina (FRB) presente en TOR. Otros dominios conservados de las proteínas TOR incluyen un largo tramo de dominios en tándem denominados HEAT (Huntingtin, EF3A, PP2A, TOR), un dominio FAT (FRAP, ATM, TRRAP), y un dominio fosfoinositida 3-kinasa asociado a quinasa. La rapamicina inhibe específicamente a TOR (mTOR) dentro del complejo mTOR 1 (mTORC1).
De hecho, cada una de estas tres cepas mutantes portaba una mutación en uno de dos genes distintos, que fueron posteriormente llamados TOR1 y TOR2. Los aminoácidos mutantes de FPR1 que confieren resistencia a la rapamicina se conservaron (es decir, también se encontraron) en FKBP12 humana y se vinculaban con las regiones compartidas de FK506 y compuestos de rapamicina, pero la pérdida de FPR1 no afectó el crecimiento de la levadura, lo que apoyó la idea de que la unión de FKBP12 es necesaria para la acción de la rapamicina, pero no es el blanco biológico que afecta la proliferación celular. Actualmente se sabe que estos objetivos son las proteínas TOR1 y TOR2 en la levadura y sus ortólogos en otras especies.
La identificación y secuenciación de los genes TOR1 y TOR2 reveló que los genes son altamente homólogos y codifican proteínas grandes con similitud con una clase de quinasas lipídicas, llamada fosfoinositida 3-quinasas o fosfoinositol 3-quinasas (PI3Ks), que habían sido descubiertas unos años antes. Estudios independientes de tres grupos, seguidos de cerca, utilizaron complejos de rapamicina - FKBP12 para purificar bioquímicamente las proteínas diana en células de mamíferos.
Cada uno de estos estudios dio como resultado la misma proteína de mamífero, con un alto grado de similaridad de secuencia respecto TOR1 y TOR2 de levaduras; fue apodada como el blanco de mamíferos de la rapamicina (mTOR). Es importante destacar que las mutantes resistentes a la rapamicina tuvieron mutaciones puntuales que afectaron un residuo altamente conservado en el dominio de unión FKBP12- rapamicina (FRB) de la proteína TOR que se requiere para la captación de rapamicina (figura 1B).
A pesar de su homología con PI3K, las proteínas TOR se han establecido como proteínas serina - treonina quinasas, que contienen un amplio rango de sustratos. Considerando que los metazoos típicamente tienen sólo una proteína TOR, la existencia de TOR1 y TOR2 en la levadura, facilitó el reconocimiento por parte de los investigadores de dos complejos TOR funcional y físicamente distintos (TORC1y TORC2), siendo solo TORC1 sensible a rapamicina. Esta es una característica conservada, con mTOR como el componente central de los complejos ortólogos en todas las células de mamíferos.
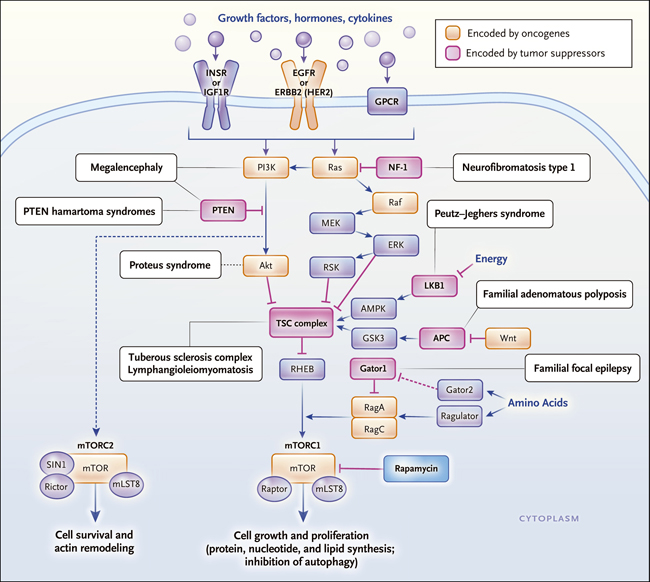
Figura 2. Muchos caminos conducen a mTORC1.
El complejo proteico mTORC1 es un punto de convergencia para múltiples vías de señalización que controlan el crecimiento y la proliferación celular. Estas son las vías detectan la presencia o ausencia de factores de crecimiento, hormonas y citoquinas a través de los receptores de la superficie celular, incluidos los receptores con actividad tirosin quinasa (por ejemplo, INSR y EGFR) y receptores acoplados a proteína G (GPCR) o nutrientes y energía intracelular (por ejemplo, aminoácidos, y ATP). Dentro de la red de señalización río arriba hay muchos oncogenes (naranja) y supresores tumorales (rosado) que frecuentemente se encuentran mutados en cánceres esporádicos o asociados con una variedad de sobrecrecimiento genético, tumores y síndromes de epilepsia (cajas blancas) caracterizados por una activación elevada de mTORC1. Se muestran los componentes esenciales de mTORC1 y mTOR 2 (mTORC2).
Desde su innovador descubrimiento, las proteínas TOR que tienen ortólogos en los cuatro reinos eucarióticos, han sido objeto de miles de estudios que han revelado la central función de las proteínas en la transmisión de señales de crecimiento desde nutrientes y factores endocrinos hasta procesos celulares subyacentes al crecimiento celular y proliferación. El desciframiento de una gran red de señalización que converge en el complejo TOR sensible a rapamicina, mTORC1, ha demostrado que está íntimamente ligado a la fisiología y patología en humanos (figura 2).
Los usos aprobados y las pruebas continuas de los inhibidores de mTOR en la clínica reflejan un aumento creciente de la sofisticada comprensión de esta red, que está formada por muchos componentes que están disfuncionales en tumores y síndromes de sobrecrecimiento, así como también en trastornos neurológicos y cánceres esporádicos. Enfermedades pleiotrópicas como el complejo de esclerosis tuberosa, que es causada por mutaciones que afectan a un regulador negativo clave de mTORC1, el complejo proteico TSC, ilustra la amplitud de las manifestaciones que pueden resultar de la señalización incontrolada de mTORC1, incluyendo tumores generalizados, epilepsia y autismo. Por último, existe un gran interés en dirigirse a mTOR para combatir enfermedades relacionadas con el envejecimiento y la edad, debido a que la inhibición de mTORC1 con rapamicina o a través de la dieta se ha encontrado que prolonga la duración de lavida útil de los organismos, desde la levadura hasta los ratones.
Fuente bibliográfica
Game of TOR — The Target of Rapamycin Rules Four Kingdoms
Brendan D. Manning, Ph.D.
Department of Genetics and Complex Diseases, Harvard T.H. Chan School of Public Health, Boston.
DOI: 10.1056/NEJMcibr1709384