Expandiendo el conocimiento sobre el genoma
Menos del 2% del genoma humano codifica proteínas. Un gran desafío para las ciencias genómicas ha sido el mapeo de los elementos funcionales - las regiones que determinan el grado de expresión de los genes - en el 98% restante de nuestro ADN. El proyecto de la Enciclopedia de Elementos de ADN (ENCODE), entre otros grandes esfuerzos de colaboración, se estableció en 2003 para crear un catálogo de estos elementos funcionales y esbozar sus actividades en la regulación de la expresión de los genes y así tener un conocimiento más acabado sobre la predisposición genética a las enfermedades. Tal es la magnitud de este proyecto, que sus resultados han sido publicados en nueve artículos en los que se entrega la tercera fase de un valioso trabajo.
Regulación en cis
En 2007, la fase piloto del proyecto ENCODE buscó elementos funcionales en el 1% del genoma de unas pocas líneas celulares humanas. El consorcio catalogó dos tipos de estos elementos. En primer lugar, identificaron regiones de ADN que se transcriben en ARN (tanto con codificación de proteínas como sin ella). En segundo lugar, identificaron regiones que regulan la transcripción de los genes, conocidas como elementos reguladores en cis (CREs). Estas regiones pueden identificarse por su accesibilidad a las enzimas que destruyen el ADN, como la DNasa I, por las proteínas de unión al ADN, como los factores de transcripción, o por modificaciones moleculares específicas en las histonas, a las que el ADN está unido en un complejo llamado cromatina.
En 2012, la segunda fase del proyecto ENCODE extendió la búsqueda de estos elementos funcionales a todo el genoma en más líneas celulares humanas, estableciendo una base sólida para la enciclopedia. Esfuerzos similares se extendieron al genoma del ratón en 2014, profundizando nuestra comprensión de estos elementos desde una perspectiva evolutiva.
En la actual y tercera fase del proyecto, el consorcio pasó de las líneas celulares a las células tomadas directamente de los tejidos humanos y de ratones, proporcionando una enciclopedia más relevante desde el punto de vista biológico. También introdujeron ensayos para investigar los aspectos más amplios de los elementos funcionales, por ejemplo, para caracterizar los elementos incorporados en los ARN o para analizar el looping de cromatina, que hace que los CREs separados se acerquen mucho entre sí para permitir la regulación de los genes (figura 1).
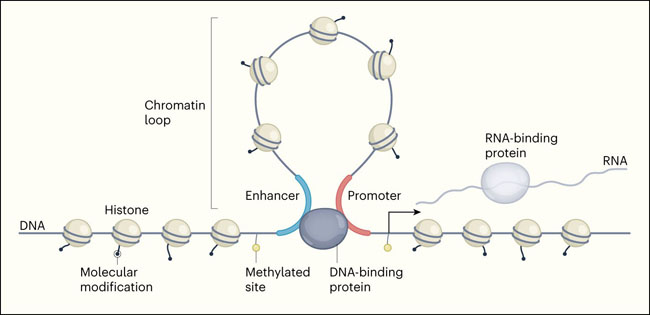
Figura 1 | Examinación de elementos funcionales a través del genoma.
Nueve artículos publicados del proyecto Enciclopedia de Elementos de ADN (ENCODE) catalogan una amplia gama de aspectos de la organización y regulación del genoma. El consorcio produjo un catálogo de elementos reguladores en cis (CREs) - secuencias de ADN llamadas promotores y potenciadores que regulan la transcripción génica (flecha negra) en sitios genómicos cercanos y lejanos, respectivamente. Los investigadores analizaron las secuencias de ADN y ARN, y las proteínas unidas, incluidas las histonas. También estudiaron las modificaciones moleculares del ADN e histonas, incluidos los sitios de metilación. Estas modificaciones pueden conducir a cambios en la regulación de los genes, y en el bucle de cromatina, lo que puede poner a los potenciadores en estrecho contacto con los genes que regulan.
En el artículo principal, el Consorcio del Proyecto ENCODE proporciona una visión amplia de conjuntos de datos recientemente añadidos de 6.000 experimentos, realizados con unas 1.300 muestras (DOI: 10.1038/s41586-020-2493-4). Mediante la integración de estos conjuntos de datos, el consorcio ha creado un registro en línea de los CREs candidatos. La mayoría están clasificados como promotores o potenciadores - CREs situados respectivamente en el sitio genómico en el que comienza la transcripción de un gen o a cierta distancia de él. El consorcio hizo un seguimiento de la actividad de cada CRE candidato, junto con las proteínas que se unen a él en muchas muestras diferentes de diversos tejidos. Utilizaron datos de bucles de cromatina para vincular los potenciadores a los genes que podrían regular. Este registro en línea marca un verdadero hito, al convertir una cantidad abrumadora de información genómica en una enciclopedia de elementos de ADN que se puede buscar, filtrar y recuperar, y que es de libre acceso en https://screen.encodeproject.org.
Los ocho artículos complementarios, profundizan en la biología que hay detrás de este proyecto. Estos estudios aprovecharon la escala y la variedad de los conjuntos de datos de ENCODE para revelar los principios que rigen el funcionamiento de los elementos funcionales. En conjunto, los trabajos demuestran el valor de la producción de datos a gran escala en la biología.
Para identificar los CREs, Meuleman y sus colegas definieron 3,6 millones de regiones de ADN accesibles en 438 tipos de células y tejidos, midiendo la susceptibilidad de todo el genoma a la DNasa I (DOI: 10.1038/s41586-020-2559-3 (2020)). Vierstra y sus colegas examinaron además los patrones de división de la DNasa I para comprender cómo se unen los factores de transcripción a los CRE (DOI: 10.1038/s41586-020-2528-x). Llegaron a la conclusión de que la mayoría de los CRE están ocupados por múltiples factores de transcripción de manera independiente y bien espaciados.
Un hallazgo sorprendente fue que cerca de la mitad de las proteínas de unión al ARN perfiladas también interactúan con el ADN. Sin embargo, los autores encontraron poca evidencia de que las proteínas se unan a ambos tipos de macromoléculas en sitios con la misma secuencia. Por lo tanto, se necesitan más estudios para evaluar si estas interacciones se producen por unión directa, están mediadas por otras proteínas de unión al ADN o están mediadas por ARN unidos a la cromatina fuera de su sitio de síntesis.
Este catálogo de elementos de ARN amplía sustancialmente nuestro conocimiento de los componentes reguladores codificados en el genoma humano. Debería permitir a los investigadores predecir las variantes genéticas que alteran el procesamiento del ARN, y constituirá un recurso inestimable para la investigación sobre cómo se regulan las interacciones proteína-ARN.
La tercera fase del proyecto ENCODE es un tour de force. Pero debido a que muchos elementos reguladores actúan solo en tipos de células específicas o en momentos particulares, no es posible evaluar con precisión la integridad de la enciclopedia. Sería interesante ver cómo el proyecto pudiera incorporar tecnologías unicelulares para desentrañar los elementos espacio temporales específicos y así desvelar aún más los fundamentos de la regulación génica. También estaríamos ansiosos por ver la convergencia del proyecto ENCODE con los esfuerzos de colaboración que tienen alcances superpuestos, por ejemplo, el Atlas de Células Humanas19 (que tiene por objeto trazar el mapa de la expresión génica en todas las células del cuerpo humano) y el proyecto del Núcleo 4D20 (que tiene por objeto comprender la organización tridimensional del cromosoma en diferentes momentos y en diversos tipos de células). La integración de los datos pertinentes de estos proyectos en la enciclopedia ampliaría aún más su cobertura.
Esta enciclopedia aún no terminada ya se ha convertido en una herramienta fundamental para comprender la regulación de los genes y la predisposición genética a las enfermedades. En la próxima cuarta fase del proyecto ENCODE, se podría ver una evaluación sistemática en relación con si los elementos reguladores en cis realizan las funciones que se infieren sobre la base de las modificaciones de las histonas y proteínas unidas; esto podría lograrse utilizando tecnologías genómicas funcionales de alto rendimiento. La continua expansión de ENCODE a contextos biológicos más amplios (como las muestras de enfermedades y los tipos de células raras) con una resolución unicelular ayudaría a los investigadores a utilizar la información genómica para diagnosticar y prevenir enfermedades.
Fuente bibliográfica
Expanded ENCODE delivers invaluable genomic encyclopedia
Chung-Chau Hon & Piero Carninci
RIKEN Center for Integrative Medical Sciences, Yokohama, Kanagawa 230-0045, Japan.
DOI: 10.1038/d41586-020-02139-1
Temas Relacionados