Edición del genoma mitocondrial, una posibilidad cercana
Las toxinas bacterianas representan un vasto reservorio de diversidad bioquímica que puede ser explotado para aplicaciones biomédicas. Entre esas moléculas figura un grupo de toxinas interbacterianas de la superfamilia de las deaminasas, cuyos miembros tienen aplicación en técnicas de edición genética. Resulta de particular interés una en particular, denominada DddA, que cataliza la desaminación de las citidinas dentro del ADN bicatenario. Esto permite la manipulación precisa del ADN mitocondrial, lo que podría tener amplias implicaciones para el estudio y posible tratamiento de los trastornos de herencia materna.
Toxina bacteriana, la clave
El ADN mitocondrial (ADNmt) codifica solo 13 proteínas, todas ellas implicadas en la generación del suministro de energía para la célula. Las mutaciones en el ADNmt pueden causar una serie de enfermedades metabólicas incurables y limitantes en los humanos. Por lo tanto, el desarrollo de herramientas para editar el genoma de las mitocondrias ha sido un objetivo largamente buscado en genética. En una reciente publicación Beverly Mok y colaboradores de la Universidad de Harvard (Cambridge, Estados Unidos) informan de un instrumento molecular que por primera vez permite la edición precisa del ADNmt. La clave de este logro fue el descubrimiento de una toxina secretada por bacterias para eliminar a sus vecinas.
La toxina bacteriana descubierta por Mok y sus colegas es una enzima citidina desaminasa llamada DddA, que cataliza la conversión del nucleótido citosina (C) en otra base, el uracilo (U). Una característica notable de la DddA es que se dirige al ADN de cadena doble, mientras que todas las citidina desaminasa previamente identificadas se dirigen al ADN monocatenario. Es crucial que, aunque los enfoques convencionales de edición del genoma implican la utilización de enzimas tipo nucleasa que actúan como tijeras moleculares para cortar el ADN de ambas cadenas, la DddA convierte la C en la U sin inducir la ruptura del ADN de doble hebra. Esto lo hace particularmente adecuado para editar el genoma mitocondrial, que carece de mecanismos eficientes para reparar las rupturas de ADN bicatenario.
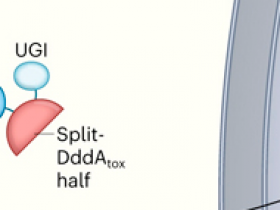
Los investigadores tuvieron que superar varios desafíos para reutilizar la DddA para la edición del genoma mitocondrial. El principal de ellos es el hecho de que la citidina desaminasa es tóxica para las células de los mamíferos. Los autores dividieron el dominio de la toxina DddA en dos partes inactivas llamadas mitades de Split-DddAtox. Fusionaron estas mitades a las proteínas TALE, que pueden ser diseñadas para unirse a secuencias específicas de ADN. La unión de los dos TALEs al ADN mitocondrial une y activa las mitades de DddAtox.
Para alcanzar el ADNmt en la matriz mitocondrial, el complejo TALE-split-DddAtox debe cruzar dos membranas mitocondriales. Por lo tanto, los científicos etiquetaron este construto con una secuencia de aminoácidos que actúa como una señal de objetivo mitocondrial. La capacidad de explotar la maquinaria existente de importación de proteínas da a este enfoque una gran ventaja sobre los sistemas guiados por ARN para la edición del genoma como el CRISPR-Cas9. Los métodos CRISPR no funcionan eficientemente en el ADNmt, posiblemente porque la célula no tiene mecanismos para importar ARN a las mitocondrias.
Otro desafío surge del hecho de que la citidina desaminasa convierte la base nitrogenada C en U, en vez de timina (T) específica del ADN. Aunque el U tiene las mismas propiedades de apareamiento de bases que la T, pertenece al ARN. La base normalmente se corta del ADN con la ayuda de una enzima llamada uracil-ADN glicosilasa y se sustituye por C.
Por lo tanto, Mok y sus coelgas fusionaron las mitades de TALE-split-DddAtox con un inhibidor de la uracilo glicosilasa (UGI). Esto protege a la U de la glicosilasa hasta que se produce la siguiente ronda de replicación o reparación del ADN, en cuyo momento la base de guanina (G) de la cadena complementaria -que se emparejó con la C antes de la edición- es sustituida por adenina (A), la base que se empareja con la T. La incorporación de UGI aumentó la eficiencia de la edición de la base de citosina unas ocho veces.
El constructo final, denominado editor de citosina derivada de DddA (DdCBE), consiste por lo tanto en una señal de objetivo mitocondrial, una proteína TALE, una mitad de DddAtox dividida y una UGI (figura 1). Mok y sus colegas demostraron que el constructo se importa eficientemente en las mitocondrias de las células humanas y puede modificar una selección de genes mitocondriales. La edición, de un par de bases C-G a T-A, ocurrió alrededor del 5 a 50% de las veces. La eficiencia de la edición fue influenciada por varios factores: el espacio entre las dos subunidades de DdCBE; el diseño del TALE; la orientación de las mitades de Split-DddAtox; y la posición de la citosina objetivo en relación con los sitios de unión del TALE.
Figura 1 | Una herramienta para corregir el genoma mitocondrial.
Mok y colaboradores han desarrollado una herramienta molecular, DdCBE, que cambia los pares de bases de citosina y guanina (C-G) a pares de timina y adenina (T-A) en el ADN de las mitocondrias. a, En el enfoque de los autores, el DdCBE se divide inicialmente en dos constructos, cada uno de los cuales contiene: una porción inactiva de una toxina bacteriana, llamada DddAtox dividida; una proteína TALE que se une a una secuencia específica de ADN mitocondrial; una secuencia de aminoácidos que dirige el constructo a la matriz mitocondrial; y una proteína inhibidora de la uracilo glicosilasa (UGI). b, La secuencia de orientación mitocondrial se pierde cuando el constructo se importa a la matriz. Las proteínas TALE se unen a las secuencias adyacentes de ADNmt, uniendo y activando así las dos mitades de Split-DddAtox. La toxina actúa como una enzima que convierte la base C objetivo en otra base, el uracilo (U). Normalmente, el U se elimina del ADN, pero la proteína UGI protege la base editada hasta que se produce la replicación del ADN (no se muestra). En este punto, es reemplazada por la T, que forma un par con la A.
Una consideración importante para todas las herramientas de edición del genoma es si modifican el ADN en sitios no deseados. Mok y sus colegas compararon las células tratadas y las no tratadas, y no encontraron ningún efecto fuera del objetivo en el genoma nuclear. Esta actividad inespecífica en el ADNmt fue baja, excepto en el caso de un gen, en el que las ediciones fuera del objetivo estaban vinculadas al diseño del TALE.
A continuación, examinaron el potencial terapéutico del DdCBE. Los autores informaron que la edición de la base de citosina tiene el potencial de corregir el 49% de las mutaciones de ADNmt perjudiciales conocidas. Sin embargo, en su forma actual, la DdCBE puede editar eficientemente solo las bases C que están precedidas en el genoma por una T, estrechando su rango.
La dependencia del DdCBE de la replicación de ADN para implementar la conversión del par C-G a T-A implica una eficiencia máxima teórica de edición del 50%. Para explicarlo, los dos ADNmt recién replicados reciben cada uno una cadena de ADN parental, una de las cuales no será editada, que contiene G, que se empareja con una C. Sin embargo, la actividad de DdCBE persiste durante varios días, lo que potencialmente ofrece la oportunidad de una mayor edición durante los eventos de replicación posteriores. El hecho de que los efectos fuera del objetivo aumenten durante la exposición prolongada a la DdCBE será una consideración clave para el futuro.
Estas advertencias significan que DdCBE podría causar una reducción, en lugar de una eliminación completa, de las mutaciones de ADNmt. Pero dado que la gravedad de los síntomas de las enfermedades mitocondriales aumenta con la carga de la mutación, la capacidad de reducir el nivel de la mutación en sí misma es prometedora desde el punto de vista terapéutico.
El trabajo de Mok y sus colegas es un avance clave para el desarrollo de terapias genéticas para las enfermedades del ADNmt. Además, se podría obtener una mejor comprensión de la relevancia de las mutaciones a nivel del genoma mitocondrial en enfermedades complejas, el cáncer y la disfunción celular relacionada con la edad. Es probable que el estudio también inspire nuevos desarrollos en la ingeniería genética y de proteínas que aumenten el alcance y eficiencia de DdCBE, junto con intensificar la búsqueda de otros candidatos a editores del genoma.
Fuente bibliográfica
Mitochondrial genome editing gets precise
Magomet Aushev & Mary Herbert
Wellcome Centre for Mitochondrial Research, Biosciences Institute, Centre for Life, Newcastle upon Tyne NE1 4EP, UK.
DOI: 10.1038/d41586-020-01974-6
Temas Relacionados