Reformulando el diagnóstico genético prenatal
Nuevo método no invasivo emplea el ADN circulante, del plasma de ambos progenitores, para detectar embarazos con un alto riesgo de trastornos genéticos mediante secuenciación profunda.
El diagnóstico genético fetal es fundamental en la atención prenatal, y los avances recientes en la secuenciación del trío-exoma prenatal (es decir, secuenciación simultánea de las regiones codificantes del genoma del feto, madre y padre) proponen beneficios diagnósticos. Sin embargo, debido a la naturaleza invasiva del muestreo fetal, su uso se reduce a fetos con anomalías estructurales identificables. Esta limitación deja muchas alteraciones monogénicas sin diagnosticar porque no se manifiestan en el período prenatal, lo que da como resultado un gran número de recién nacidos con trastornos pediátricos letales y gravemente debilitantes.
El desarrollo de pruebas fetales no invasivas que utilizan ADN circulante de una muestra de sangre materna ha revolucionado el cribado prenatal, pero su aplicación se ha limitado principalmente a las alteraciones cromosómicas debido a la baja resolución de las pruebas existentes.
Para abordar estas limitaciones, desarrollaron un examen prenatal no invasivo mediante secuenciación profunda del trío-exoma. Este método integral tiene una alta sensibilidad para detectar variantes fetales de un solo nucleótido, pequeñas inserciones y deleciones, mutaciones con un gran número de copias y aneuploidías cromosómicas a partir de ADN circulante obtenido de una muestra de sangre materna (figura 1). Este enfoque de prueba de concepto aprovecha la secuenciación ultra profunda del trío-exoma con corrección de errores, para detectar variantes fetales de novo (mutaciones nuevas no heredadas) con alta precisión. Estas son la causa principal de trastornos graves del desarrollo pediátrico.
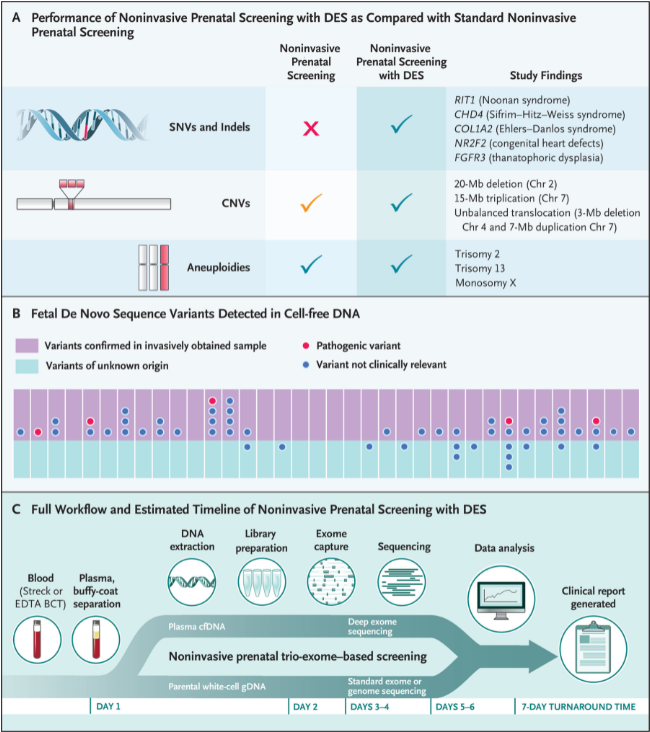
Figura 1: rendimiento y flujo de trabajo del cribado prenatal no invasivo con secuenciación profunda del trío-exoma.
En este estudio, reclutaron a 36 mujeres embarazadas y sus parejas. Los criterios de inclusión clave fueron una medición de la translucencia nucal de mínimo 5 mm, al menos una anomalía fetal identificada durante el primer o segundo trimestre gestacional, o ambos. Realizaron una secuenciación ultra profunda del exoma en el ADN libre extraído del plasma materno, logrando una gran cobertura de secuenciación por muestra. Se utilizó un proceso personalizado para nombrar y filtrar variantes con precisión, incorporando corrección de errores mediante identificadores moleculares únicos, modelado de ruido específico del sitio y estimación de la fracción fetal que osciló entre 3,72-19%.
Se realizó un análisis de trío-exoma, incluyendo el plasma materno y muestras de sangre parental compatibles, para la identificación de variantes fetales nuevas y paternas heredadas. El rendimiento de la detección prenatal no invasivo utilizando la secuenciación profunda del trío-exoma se evaluó mediante los resultados obtenidos con este nuevo método, en comparación con la secuenciación del exoma completo de forma invasiva, la del genoma completo y el análisis de microarrays cromosómicos de las vellosidades coriónicas o muestras del líquido amniótico, para todos los casos.
Se identificó las siguientes variantes patogénicas fetales de novo, que se consideran clínicamente relevantes en el embarazo: cuatro mutaciones de un solo nucleótido, una pequeña deleción, dos trisomías autosómicas, una aneuploidía de un cromosoma sexual, dos variantes con un gran número de copias y una translocación desequilibrada. Todas las mutaciones que se detectaron con este método no invasivo se confirmaron en las muestras fetales obtenidas de forma invasiva. Estas se alinearon con el fenotipo fetal y proporcionaron un diagnóstico genético en 11 de los 36 embarazos. No se identificaron variantes adicionales mediante los análisis invasivos en los 36 casos. Por lo tanto, se observa una concordancia total entre el análisis prenatal no invasivo y el invasivo, con una detección del 100% de variantes patógenas de novo.
Para evaluar la representación del exoma fetal en el ADN circulante, ampliaron el análisis para abarcar todas las variantes de secuencia fetal heredadas por vía paterna dentro de la región codificante. La sensibilidad media del nuevo método para la detección de estas mutaciones en todos los genes codificantes de proteínas fue del 95,12%.
En un subconjunto de muestras de plasma, esta nueva técnica reveló un pequeño número de variantes de origen desconocido, que no se identificaron en las correspondientes muestras obtenidas de forma invasiva. Sin embargo, ninguna de estas se consideró clínicamente relevante o requirió más pruebas.
En este método no invasivo se incluyeron muestras de los padres, lo que permite el tamizaje de portadores en ambos progenitores para identificar embarazos que tienen un alto riesgo de sufrir trastornos recesivos. Este enfoque de prueba demuestra que, puede determinar con precisión la herencia fetal de mutaciones paternas en el caso de que la madre no sea portadora de las mismas. La confirmación de la presencia de una variante heredada de la madre en el feto requeriría pruebas invasivas.
Este tipo de cribado prenatal proporciona un medio para detectar, de forma no invasiva y exhaustiva, una amplia gama de trastornos genéticos sin suponer un riesgo para el feto o la madre. La integración de esta técnica en la atención prenatal de rutina junto con el cribado ecográfico brindaría una oportunidad para mejorar las tasas de detección temprana, reducir el número de procedimientos invasivos y facilitar las intervenciones rápidas. Aunque se necesitan grandes estudios para probar esta hipótesis, los hallazgos de esta investigación (figura 1B) predecirían una tasa baja de falsos positivos. Todos los resultados positivos deben confirmarse mediante pruebas de diagnóstico invasivas prenatales. Con un tiempo de respuesta de aproximadamente 7 días (figura 1C), la detección prenatal no invasiva mediante secuenciación profunda del trío-exoma proporciona resultados oportunos.
Los hallazgos respaldan una mayor investigación de la secuenciación profunda del exoma del ADN circulante para detectar variantes patogénicas en fetos con y sin anomalías estructurales identificables.
Fuente bibliográfica
Comprehensive Noninvasive Fetal Screening by Deep Trio-Exome Sequencing
Ieva Miceikaitė, et al.
University of Southern Denmark, Odense, Denmark
N Engl J Med 2023; 389:2017-2019